Leere Seite
Seite noch im Aufbau!
Einstieg/Wiederholung
Zur Erinnerung: Das langfristige Ziel dieser Einheit ist es, zu klären, wie ein Stück DNS die Ausprägung eines Merkmals (also z.B. eure Haarfarbe) beeinflussen kann.
In der letzten Einheit habt ihr den Aufbau der DNS kennengelernt. Bevor wir uns anschauen, wie dieser Aufbau nun ein sichtbares Merkmal beeinflussen kann, zunächst ein Zwischenschritt:
Euer aktueller Körper, in dem ihr steckt, besteht aus ca. 100 000 000 000 000 Zellen. Entstanden seid ihr aber alle zunächst aus einer einzigen befruchteten Eizelle. Das Erbgut in dieser befruchteten Eizelle musste sehr oft vervielfältigt werden, damit alle Zellen eures heutigen Körpers exakt das gleiche Erbgut enthalten und eine Einheit bilden. Ihr kennt den Prozess schon, der hierbei eine entscheidende Rolle spielt: Die Mitose.
Sucht das Arbeitsblatt zum Thema Mitose heraus (auch im Buch wäre eine Abbildung)! Analysiert die einzelnen Schritte noch einmal und formuliert dann einen Satz, der die relativ simple Frage beantwortet:
- Was passiert in der Meiose (weniger auf den Mechanismus eingehen, sondern auf das Ergebnis)?
Die Meiose ist ein Abschnitt während der Zellteilung, bei der das Erbgut eines Zellkerns in zwei gleich große Portionen aufgeteilt wird, die jeweils in den beiden entstehenden Tochterzellen landen.
An dem oben stehenden Satz erkennt man, dass folgende Aspekte berücksichtigt wurden (überprüft, ob euch das klar ist!):
- Die Meiose ist nur ein Abschnitt der Zellteilung
- Bei der Meiose wird das Erbgut im Zellkern halbiert
- Die Tochterzellen enthalten nach der Meiose zunächst nur die Hälfte des ursprünglichen Erbguts.
Vorüberlegungen 1
Die Tochterzellen einer Zelle, die sich soeben geteilt hat, besitzen also nur die Hälfte des "normalen" Erbguts. Die folgende Abbildung verdeutlicht die Situation noch einmal an einem Chromosom. Macht euch anhand dieser Abbildung noch einmal klar: Aus wie vielen DNS-Fäden besteht das Erbgut eines Menschen? Ohne numerische Chromosomenaberration! (was war das noch mal?)
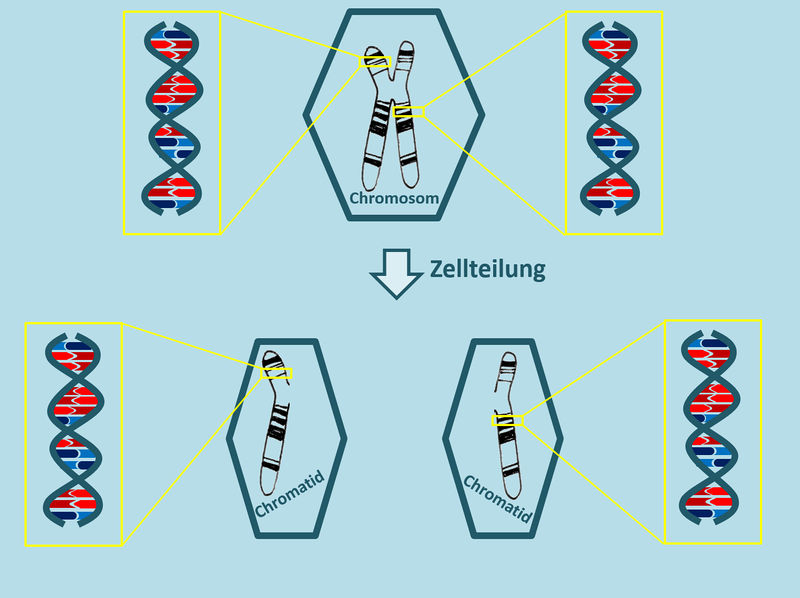
- Numerische Chromosomenaberration: Abweichung von der "normalen Anzahl" an Chromosomen. Bsp.: Trisomie-21, Turner-Syndrom, Klinefelter-Syndrom
- Normalerweise: 23 Chromosomenpaare entspricht 46 einzelnen Chromosomen entspricht 92 Chromatiden. Ein Chromatid entspricht einem DNS-Faden, also besteht das Erbgut eines Menschen normalerweise aus 92 DNS-Fäden.
Vorüberlegungen 2
Damit sich eine Tochterzelle erneut teilen kann, muss das "halbe Erbgut" zunächst wieder verdoppelt werden. Dieser Prozess nennt sich Replikation. Die Forscher, die maßgeblich an der Entschlüsselung dieses Prozesses mitgewirkt haben, waren Matthew Meselson und das Ehepaar Mary und Frank Stahl (müssten alle noch leben). Das Meselson-Stahl-Experiment soll hier nachempfunden werden.
Rein theoretisch gibt es drei verschiedene Möglichkeiten wie ein DNS-Faden repliziert (verdoppelt) werden kann:
- konservativ: Könnte man mit einem Kopierer vergleichen. Es gibt ein Orginal, das unverändert bleibt und ein Duplikat, das aus neuen Bausteinen zusammengebastelt wird.
- semikonservativ: Bei der DNS sind die beiden Einzelsträng komplementär. Das bedeutet, wenn ich den einen Einzelstrang kenne, kann ich den fehlenden einfach erzeugen. Das ermöglich eine semikonservative Replikation: Die doppelsträngige DNS wird in ihre zwei Einzelstränge getrennt, und es wird jeweils ein neuer ergänzt.
- dispers: Das könnte man wohl am ehesten mit... "irgendwie" oder "durcheinander" übersetzen. Gemeint ist: Das Original wird zerstückelt und mit neuen Bausteinen zu zwei Abbildungen des Originals wieder zusammengesetzt. Dafür gibt es in unserer Lebensumwelt kein vernünftiges Beispiel.
Die folgenden Abbildungen zeigen einen grafischen Überblick über diese drei Varianten:
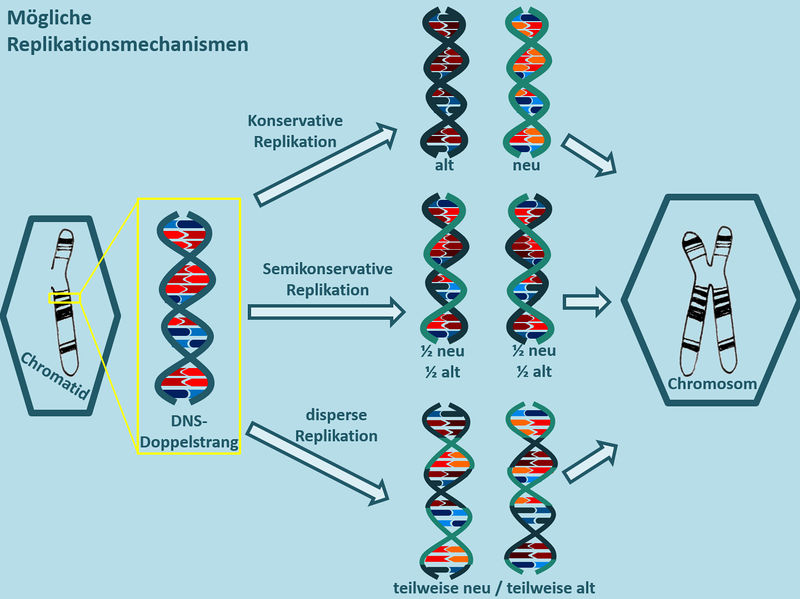
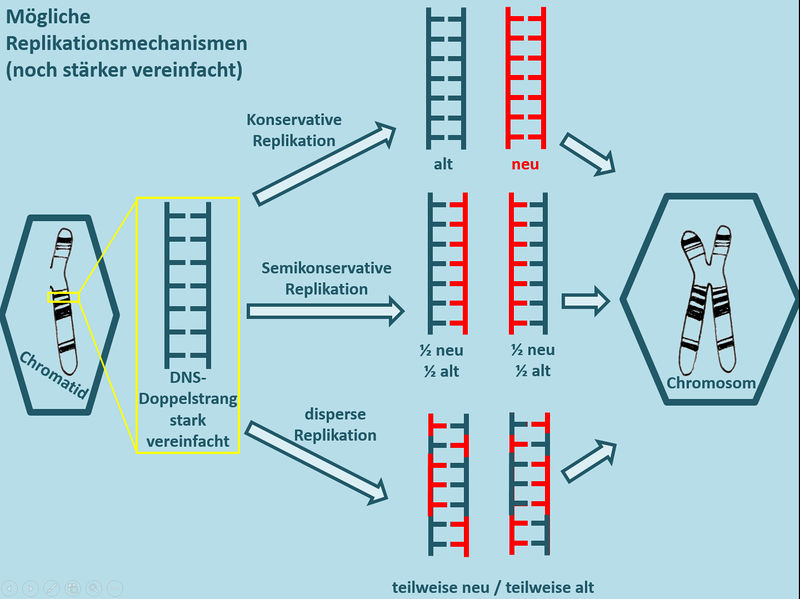
Das Problem an dieser Darstellung: Auf den Bildern könnt ihr aufgrund von Farben sehr schön sehen, welcher Teil der DNS alt ist und welcher neu. In der Realität geht das nicht! Erstens gibt es überhaupt kein Mikroskop, mit dem man einen DNS-Strang überhaupt sehen könnte und selbst wenn, wüsste man nicht, was an einem DNS-Strang alt und was neu ist...
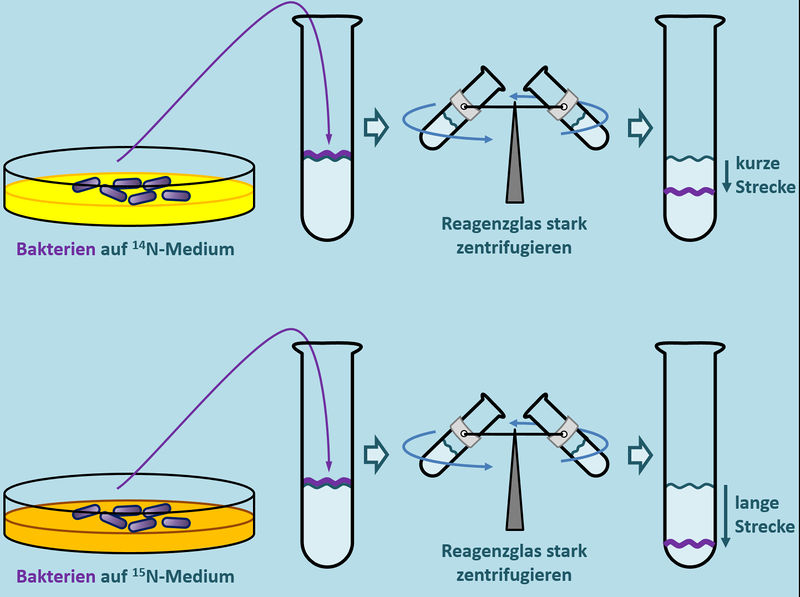
Wir haben schon oft über solche Phänomene gesprochen: Man braucht ein Experiment, mit dem man etwas sichtbar machen kann, was eigentlich nicht sichtbar ist. Und genau hier kommt das Experiment von Meselson und Stahl ins Spiel. Sie machten sich folgenden Effekt zu Nutze: Isoliert man DNS aus Bakterien, kann man sie in einem Reagenzglas auf eine Salz-Lösung auftragen und den Ansatz stark zentrifugieren. Je nachdem, wie "schwer" (das ist nicht ganz korrekt, aber ich bleibe mal bei diesem Begriff) die DNS ist, wird sie beim Zentrifugieren durch die Zentrifugalkraft im Reagenzglas weiter nach unten gedrückt (oder gezogen, wie ihr wollt).
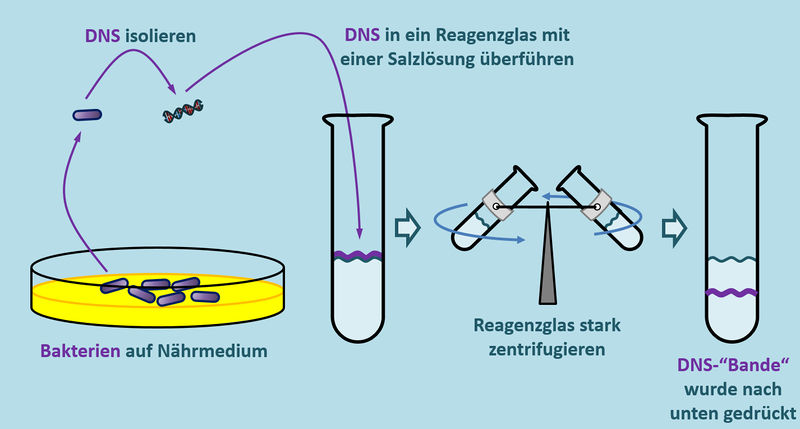
Wenn man das Experiment mehrfach wiederholt, kommt logischerweise immer das gleiche Ergebnis heraus: Die isolierte DNS wandert immer die gleiche Strecke im Reagenzglas nach unten. Meselson und Stahl haben nun aber einen Weg gefunden, die DNS in den Bakterien zu manipulieren. Sie konnten sie schwerer machen als normal: Sie ließen die Bakterien auf einem Medium wachsen und sich vermehren, welches schwere Stickstoff-Atome enthielt (man symbolisiert schweren Stickstoff mit 15N). Auch die DNS enthält Stickstoff-Atome. Normalerweise leichten Stickstoff (14N), weil nur der in der Natur in großen Mengen vorkommt. Nachdem den Bakterien im Versuch aber nur schwerer Sticktstoff 15N zur Verfügung stand, mussten sie diesen zum Aufbau ihrer DNS heranziehen. Lässt man die Bakterien lange genug in diesem 15N-Medium wachsen, enthalten sie nach einiger Zeit nur noch "schwere DNS". Zentrifugiert man nun die DNS von diesen Bakterien, stellt man tatsächlich fest, dass diese DNS etwas weiter nach unten gedrückt/gezogen wird als die leichte DNS der ursprünglichen Bakterien.
Was bringt das jetzt?
Noch gar nichts! Man kann erstmal nur unterscheiden, ob die DNS schwer oder leicht ist. Aber der Versuch war noch nicht zu Ende. Meselson und Stahl überführten nun Bakterien, die ihr Leben lang auf Medium mit schwerem Stickstoff 15N gewachsen waren, auf ein neues Medium, das nur leichten Stickstoff 14N enthielt. Dort durften die Bakterien genau so lange bleiben, bis sie sich einmal geteilt hatten, das Erbgut also genau einmal verdoppelt worden war.
Anschließend wurde die DNS wieder isoliert und zentrifugiert.
Welches Ergebnis sollte man erhalten, bei:
- einer konservativen,
- einer semikonservativen und
- einer dispersen
Replikation?
- konservativ: Es sollte eine Bande mit schwerer und eine Bande mit leichter DNS auftauchen. Nachdem beim konservativen Mechanismus die alte (schwere) DNS erhalten bleiben würde und eine neue (leichte) DNS erzeugt werden würde, müsste man diese beiden Banden finden.
- semikonservativ: Es sollte nur eine Bande auftauchen; und zwar zwischen der Stelle, an der normalerweise die schwere DNS auftauchen würde und der der Stelle, an der normalerweise die leichte DNS auftauchen würde. Beim semikonservativen Mechanismus wird die alte (schwere) DNS in der Mitte geteilt und jeweils eine Hälfte durch neue Bausteine ergänzt. Das bedeutet, alle DNS-Stränge sind gleich (halb schwer, halb leicht, also: mittelschwer).
- dispers: Nachdem hier alle möglichen Varianten denkbar sind, sollten viele verschiedene DNS-Varianten auftauchen. Es sollte also keine klare Bande entstehen, sondern eher ein verschwommener Fleck
Das tatsächliche Ergebnis seht ihr hier:
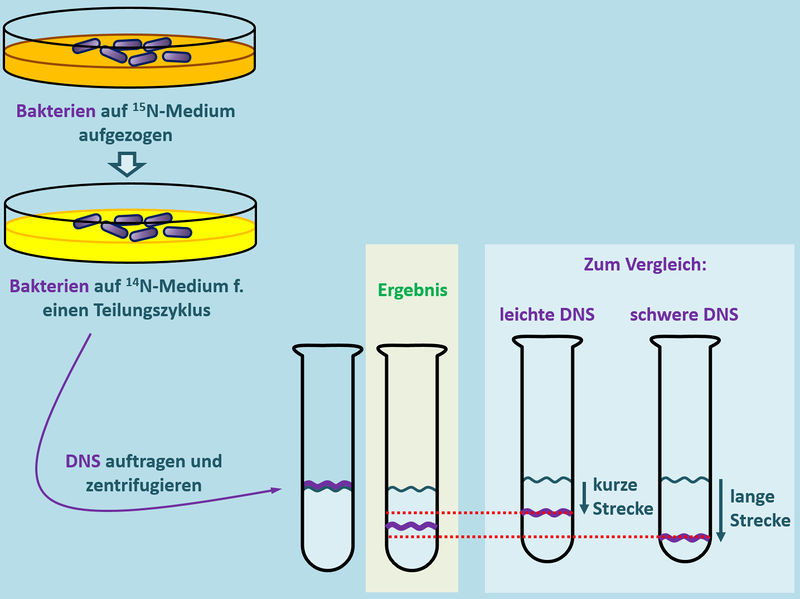
Welcher Replikationsmechanismus liegt also vor?
Der semikonservative
Wie funktioniert´s?
Schaut das Video, welches zeigt, wie die Replikation auf molekularer Ebene abläuft! Beantwortet während des Videos bzw. danach folgende Fragen:
- Was macht die Helicase?
- Wie heißt das Enzym, das aus RNA-Stückchen einen kleinen Primer formt?
- Warum kann der "leading-strand" (Vorwärts-Strang) in einem Stück ergänzt werden, der "lagging-strand" (Rückwärts-Strang) nicht?
- Was ist ein Okazaki-Fragment?
- Was macht die Ligase?
- Sie entdrillt die DNS-Doppelhelix und trennt die beiden Einzelstränge voneinander.
- Primase
- Weil die Polymerase nur in eine Richtung arbeiten kann (am neu entstehenden Strang die Nukleotide von 5´ nach 3´ verknüpfen), auf dem Vorwärtsstrang kann die Polymerase daher der Helicase "hinterher" laufen, auf dem Rückwärtsstrang muss gewartet werden, bis ein neuer Primer erstellt wurde, erst dann kann die Polymerase "von der der Helicase weg" arbeiten.
- Die noch nicht miteinander verknüpften doppelsträngigen Abschnitte auf dem Rückwärtsstrang
- Sie schließt die Lücke im Zucker-Phosphat-Gerüst der neu synthetisierten Einzelstränge.
Hausaufgabe
Lesen: Buch, S. 63 - 65 (ohne den blauen Zettelkasten)
Aufgabe: Beschreibe das sichtbare Ergebnis, wenn man die Bakterien im Versuch von Meselson und Stahl nicht einen Teilungszyklus lang, sondern zwei Teilungszyklen lang auf Medium mit leichtem Stickstoff wachsen lässt!

