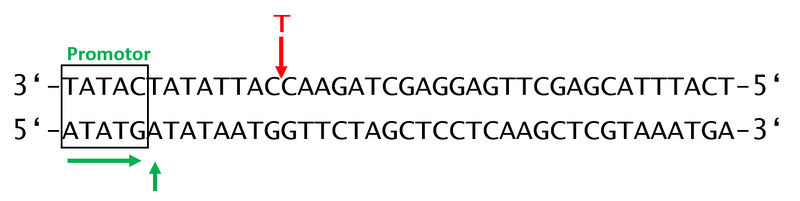Leere Seite: Unterschied zwischen den Versionen
| (15 dazwischenliegende Versionen von einem Benutzer werden nicht angezeigt) | |||
| Zeile 1: | Zeile 1: | ||
<span style="color:#F00">'''Seite noch im Aufbau!'''</span> | <span style="color:#F00">'''Seite noch im Aufbau!'''</span> | ||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
<div style="margin:0; margin-right:8px; border:0px solid #dfdfdf; padding: 0em 1em 1em 1em; background-color:#DFF; align:left;"> | <div style="margin:0; margin-right:8px; border:0px solid #dfdfdf; padding: 0em 1em 1em 1em; background-color:#DFF; align:left;"> | ||
| − | <span style="color:# | + | <span style="color:#006">'''Aufgabe'''</span><br> |
| − | + | Zunächst eine Aufgabe, die mit dem letzten Thema "Mutationen" zusammenhängt: <br> | |
| + | * Ein Mutationstyp wurde im Unterricht nicht besprochen: Die so genannte '''Rastermutation'''. | ||
| + | * Bei einer Rastermutation wir eine Base (bzw. mehrere) zusätzlich in die DNA eingefügt (= '''Insertion''') oder eine Base (bzw. mehrere) entfernt (= '''Deletion'''). | ||
| + | * Dadurch wird ab diesem Punkt das gesamte '''Leseraster''' der DNA verschoben. Es kommt zur Bildung völlig anderer AS-Ketten, die so gut wie nie die Funktion des ursprünglichen Proteins erfüllen können. | ||
| + | * s. auch Buch: S. 76 letzter Absatz - S. 77 erster Absatz | ||
| + | * ein Beispiel für einen chemischen Stoff, der in der Lage ist, eine Rastermutation zu verursachen, wäre z.B. [https://de.wikipedia.org/wiki/Ethidiumbromid Ethidiumbromid] | ||
<br> | <br> | ||
| − | [[Datei: | + | Eine Beispielaufgabe: |
| − | + | * Betrachte den folgenden DNA-Strang. Zunächst nur den mit schwarz dargestellten Normalfall: <br> | |
| − | * | + | [[Datei:GenMut_Raster_AA.jpg|800px]] |
| − | + | * Leite den entsprechenden mRNA-Strang ab und übersetze diesen in eine AS-Kette (Code-Sonne auf S. 68 im Buch) | |
| − | + | ||
| − | + | ||
| − | + | ||
| − | + | ||
| − | + | ||
| − | + | ||
{{versteckt| | {{versteckt| | ||
| − | [[Datei: | + | [[Datei:GenMut_Raster_ML_T1.jpg|800px]] <br> |
| − | + | Typische Fehler: | |
| − | + | * falschen Strang abgelesen | |
| − | * | + | * Translation nicht bei AUG begonnen |
| − | * | + | |
}} | }} | ||
| − | |||
<br> | <br> | ||
| − | + | * Füge nun - wie in rot dargestellt - an der gekennzeichneten Position das Nukleotid mit der Base Adenin ein und führe erneute eine Transkription und Translation durch! | |
| − | + | ||
| − | + | ||
| − | + | ||
| − | + | ||
| − | + | ||
| − | + | ||
{{versteckt| | {{versteckt| | ||
| − | + | [[Datei:GenMut_Raster_ML_T2.jpg|800px]] <br> | |
}} | }} | ||
| − | + | * Beschreibe (am besten schriftlich, damit Du das Formulieren übst) welche Konsequenzen diese Mutation für das Lebewesen hat! | |
| − | + | ||
| − | + | ||
| − | + | ||
| − | + | ||
| − | * | + | |
| − | + | ||
| − | + | ||
{{versteckt| | {{versteckt| | ||
| − | + | Das entstehende Protein besteht aus völlig anderen Aminosäuren. Die 3dimensionale Raumstruktur wird sich völlig ändern. Da ein wichtiger Zusammenhang zwischen dieser Struktur und der Funktion einen Proteins besteht, ist das Produkt dieser Proteinbiosynthese höchstwahrscheinlich komplett funktionslos. Handelt es sich z.B. um ein Enzym, sind schwerwiegende Stoffwechselstörungen im Organismus zu erwarten. | |
| − | + | ||
}} | }} | ||
| + | </div> | ||
<br> | <br> | ||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | <div style="margin:0; margin-right:8px; border:0px solid #dfdfdf; padding: 0em 1em 1em 1em; background-color:#DFC; align:left;"> | |
| − | + | <span style="color:#060">'''Neu: Gentechnische Werkzeuge und Verfahren - Überblick'''</span><br> | |
| − | + | '''Ziel''': Der Mensch ist inzwischen in der Lage, das Erbgut von Lebewesen gezielt zu verändern. Damit kann man z.B. | |
| − | + | * die Eigenschaften von Pflanzen verändern, | |
| − | + | * Bakterien und Hefen dazu veranlassen, Stoffe in großen Mengen herzustellen, die der Mensch dann isolieren und weiterverwenden kann, | |
| − | * | + | * genetische "Defekte" zu "reparieren". |
| − | + | ||
<br> | <br> | ||
| + | Es gibt einen inzwischen etwas in die Jahre gekommenen "Selbstlernkurs", den ich die Schülerinnen und Schüler meiner Bio-Oberstufenkurse als Einleitung zur Thematik im Computerraum immer alleine bearbeiten habe lassen. Das klappte eigentlich immer ganz gut. Der Kurs wurde von einem Herrn Mallig in Freiburg entwickelt. | ||
| + | * Für diesen Selbstlernkurs solltet ihr euch ca. 30-45 Minuten Zeit nehmen. | ||
| + | * Evtl. ist es für den/die eine/n oder andere/n für euch besser '''vor dem Selbstlernkurs''' die '''Seiten im Buch''' zu lesen und einen '''kurzen Film''' zu schauen. Springt dazu zunächst zum nächsten Kasten "Weiteres Material". | ||
| + | * Der folgende Link führt euch zur Startseite [http://www.mallig.eduvinet.de/bio/gentecnk/gentek10.htm Selbstlernkurs-Start] | ||
| + | : Dort wird noch mal erklärt, was ein Selbstlernkurs ist und man steigt in die Thematik "Gentechnik" ein. | ||
| + | : Ihr kommt immer zur nächsten Seite mit einem recht unscheinbaren '''Link unten rechts''' auf jeder Seite "zur nächsten Seite". | ||
| + | : Zurück kommt ihr am besten mit den "Back"-Buttons eures Browsers | ||
| + | * Solltet ihr euch im Netz des Selbstlernkurses verlieren, könnt ihr auch immer wieder auf der folgenden Seite einsteigen: [http://www.mallig.eduvinet.de/bio/gentecnk/gentek12.htm Selbstlernkurs-Übersicht] | ||
| + | : Denn hier sind genau die Begriffe aufgeführt, die ihr beherrschen sollt! | ||
| + | : Ihr sollt erklären können: | ||
| + | :: Was "können" '''Restriktionsenzyme'''? | ||
| + | :: Was "können" '''Ligasen'''? | ||
| + | :: Was sind '''Vektoren'''? | ||
| + | :: Was bedeutet '''Klonierung'''? | ||
| + | :: Wie funktioniert die '''PCR (Polymerase-Chain-Reaction)'''? | ||
| + | :: Was ist '''cDNA'''? | ||
</div> | </div> | ||
<br> | <br> | ||
<div style="margin:0; margin-right:8px; border:0px solid #dfdfdf; padding: 0em 1em 1em 1em; background-color:#DFC; align:left;"> | <div style="margin:0; margin-right:8px; border:0px solid #dfdfdf; padding: 0em 1em 1em 1em; background-color:#DFC; align:left;"> | ||
| − | <span style="color:#060">''' | + | <span style="color:#060">'''Weiteres Material'''</span><br> |
| − | + | '''Film''' auf BRalpha, ca. 15min.: Gesamtüberblick "Was kann Gen-Technik" | |
| − | {{#ev:youtube | | + | {{#ev:youtube |jc_iY5fnGLg}}<br> |
| − | + | ||
| − | + | * Buch, S. 112-113 (Restriktionsenzyme, Ligasen, Marker) | |
| + | * Buch, S. 114-115 (Vektoren) | ||
| + | * Buch, S. 118-119 (PCR) | ||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
</div> | </div> | ||
| − | |||
Aktuelle Version vom 19. Juni 2020, 19:37 Uhr
Seite noch im Aufbau!
Aufgabe
Zunächst eine Aufgabe, die mit dem letzten Thema "Mutationen" zusammenhängt:
- Ein Mutationstyp wurde im Unterricht nicht besprochen: Die so genannte Rastermutation.
- Bei einer Rastermutation wir eine Base (bzw. mehrere) zusätzlich in die DNA eingefügt (= Insertion) oder eine Base (bzw. mehrere) entfernt (= Deletion).
- Dadurch wird ab diesem Punkt das gesamte Leseraster der DNA verschoben. Es kommt zur Bildung völlig anderer AS-Ketten, die so gut wie nie die Funktion des ursprünglichen Proteins erfüllen können.
- s. auch Buch: S. 76 letzter Absatz - S. 77 erster Absatz
- ein Beispiel für einen chemischen Stoff, der in der Lage ist, eine Rastermutation zu verursachen, wäre z.B. Ethidiumbromid
Eine Beispielaufgabe:
- Betrachte den folgenden DNA-Strang. Zunächst nur den mit schwarz dargestellten Normalfall:
- Leite den entsprechenden mRNA-Strang ab und übersetze diesen in eine AS-Kette (Code-Sonne auf S. 68 im Buch)
- Füge nun - wie in rot dargestellt - an der gekennzeichneten Position das Nukleotid mit der Base Adenin ein und führe erneute eine Transkription und Translation durch!
- Beschreibe (am besten schriftlich, damit Du das Formulieren übst) welche Konsequenzen diese Mutation für das Lebewesen hat!
Das entstehende Protein besteht aus völlig anderen Aminosäuren. Die 3dimensionale Raumstruktur wird sich völlig ändern. Da ein wichtiger Zusammenhang zwischen dieser Struktur und der Funktion einen Proteins besteht, ist das Produkt dieser Proteinbiosynthese höchstwahrscheinlich komplett funktionslos. Handelt es sich z.B. um ein Enzym, sind schwerwiegende Stoffwechselstörungen im Organismus zu erwarten.
Neu: Gentechnische Werkzeuge und Verfahren - Überblick
Ziel: Der Mensch ist inzwischen in der Lage, das Erbgut von Lebewesen gezielt zu verändern. Damit kann man z.B.
- die Eigenschaften von Pflanzen verändern,
- Bakterien und Hefen dazu veranlassen, Stoffe in großen Mengen herzustellen, die der Mensch dann isolieren und weiterverwenden kann,
- genetische "Defekte" zu "reparieren".
Es gibt einen inzwischen etwas in die Jahre gekommenen "Selbstlernkurs", den ich die Schülerinnen und Schüler meiner Bio-Oberstufenkurse als Einleitung zur Thematik im Computerraum immer alleine bearbeiten habe lassen. Das klappte eigentlich immer ganz gut. Der Kurs wurde von einem Herrn Mallig in Freiburg entwickelt.
- Für diesen Selbstlernkurs solltet ihr euch ca. 30-45 Minuten Zeit nehmen.
- Evtl. ist es für den/die eine/n oder andere/n für euch besser vor dem Selbstlernkurs die Seiten im Buch zu lesen und einen kurzen Film zu schauen. Springt dazu zunächst zum nächsten Kasten "Weiteres Material".
- Der folgende Link führt euch zur Startseite Selbstlernkurs-Start
- Dort wird noch mal erklärt, was ein Selbstlernkurs ist und man steigt in die Thematik "Gentechnik" ein.
- Ihr kommt immer zur nächsten Seite mit einem recht unscheinbaren Link unten rechts auf jeder Seite "zur nächsten Seite".
- Zurück kommt ihr am besten mit den "Back"-Buttons eures Browsers
- Solltet ihr euch im Netz des Selbstlernkurses verlieren, könnt ihr auch immer wieder auf der folgenden Seite einsteigen: Selbstlernkurs-Übersicht
- Denn hier sind genau die Begriffe aufgeführt, die ihr beherrschen sollt!
- Ihr sollt erklären können:
- Was "können" Restriktionsenzyme?
- Was "können" Ligasen?
- Was sind Vektoren?
- Was bedeutet Klonierung?
- Wie funktioniert die PCR (Polymerase-Chain-Reaction)?
- Was ist cDNA?
Weiteres Material
Film auf BRalpha, ca. 15min.: Gesamtüberblick "Was kann Gen-Technik"
- Buch, S. 112-113 (Restriktionsenzyme, Ligasen, Marker)
- Buch, S. 114-115 (Vektoren)
- Buch, S. 118-119 (PCR)